环境DNA(eDNA)方法能高效地检测水生生物多样性已逐渐成为共识,然而能否用水体eDNA对陆生生物进行广泛调查尚未有定论。近日,北京大学生态研究中心和北京大学生命科学学院姚蒙课题组在Journal of Environmental Management杂志在线发表了题为“Urban landscape-level biodiversity assessments of aquatic and terrestrial vertebrates by environmental DNA metabarcoding”的研究论文。本研究采集了北京109个静、动水水体的环境DNA(eDNA)样品,利用单一一对宏条形码引物开展了多脊椎动物类群的生物多样性调查,获得了北京地区的鱼类、爬行类、鸟类和哺乳类等多个脊椎动物类群的物种多样性分布数据,评估了eDNA方法对于不同动物类群的检测效率,并比较了静、动水水体在基于eDNA方法生物多样性监测中的差异表现。研究显示水体eDNA检测方法可以实现对水生和陆生生物的大规模系统调查,帮助发现生物多样性分布热点地区,助力城市及其他生态系统的保护和管理。
通过采集北京地区109个水体(图1)的水环境样品提取eDNA,并利用12S区段的宏条形码引物Tele02进行扩增和高通量测序,共鉴定获得脊椎动物MOTU(分子可操作分类单元)126个,包括73种鱼类,39种鸟类,11种哺乳类和3种爬行类(图2)。其中93个为本地物种,33个为非本地物种。
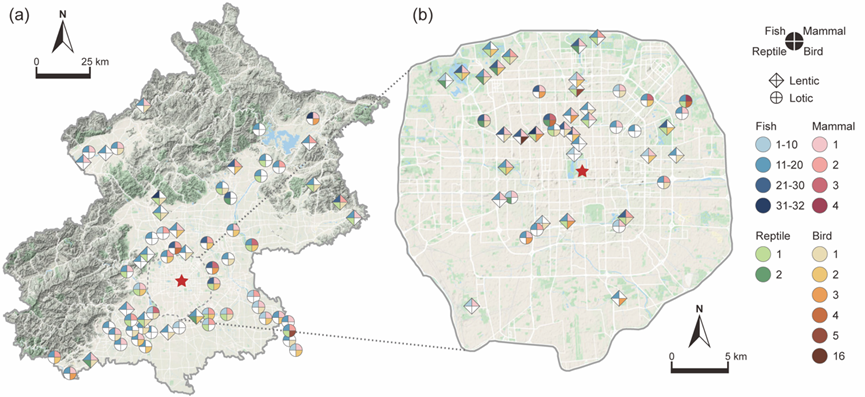
图1 北京地区脊椎动物多样性调查采样地图(a)北京市地图(b)北京五环内地图及各采样点检出的不同动物类群的丰富度。
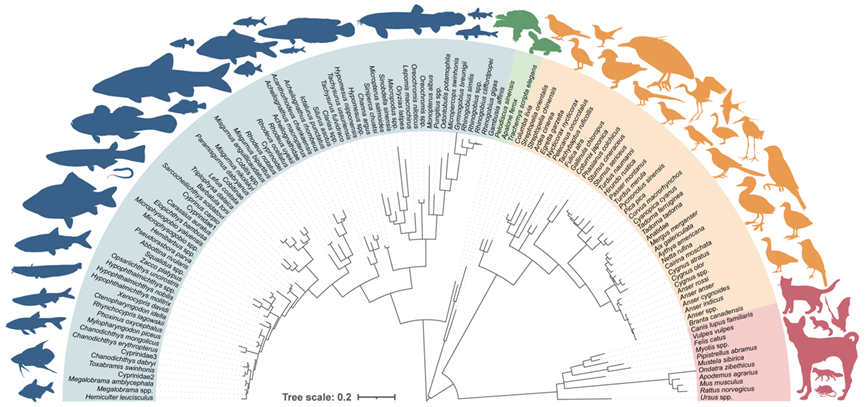

图2 基于eDNA方法检出的北京地区126种脊椎动物的系统发育树及部分检出的动物(物种图片来源于网络)
对各生物类群的物种丰富度进行物种累积曲线分析,结果显示鱼类物种丰富度随采样点数量增加的增度最快,其次为鸟类、哺乳类和爬行类(图2a);随采样点数量的增加,静水水体检出的鸟类物种相比于动水水体增长速率更快(图2c)。此外,物种累积曲线的外推曲线表明,通过增加采样点数量可以检测到更多的鸟类和哺乳类(图3c-d),这表明加大采样力度可以提高对鸟类和哺乳类多样性的检测覆盖度。此外,在静水水体中所有脊椎动物和鸟类的检测概率均显著高于动水水体,并且静水水体中检出的鱼类多样性与水体面积呈显著正相关。这可能与静水水体流速缓慢且水体更新受限导致的eDNA积累有关。

图3 随采样点增加eDNA方法对不同脊椎动物类群丰富度检测的累积曲线。(a) 各类群在全部调查水体采样点的累积曲线;(b)-(e)分别显示鱼类、鸟类、哺乳类和爬行类在不同水体类型采样点的累积曲线。Lentic,静水;Lotic,动水。
将每个水体采集的3份环境水样品作为3次采样事件来评估每个MOTU的检测概率。结果显示,基于eDNA方法的检测概率在不同物种以及不同生物类群间均存在差异,水鸟的检测概率显著高于林鸟(图4);且鱼类的检测概率显著高于鸟类和哺乳类(图5)。研究结果表明,各生物类群的生活方式会影响基于eDNA的生物多样性检测,eDNA对鱼类的检测能力高于陆生动物(鸟类和哺乳类),但研究区域常见的鸟类和哺乳类表现出与常见鱼类相似的高检测概率,这表明eDNA方法对不同脊椎动物类群的常见物种具有相当的检测能力。

图4 基于eDNA方法的不同脊椎动物物种检测概率
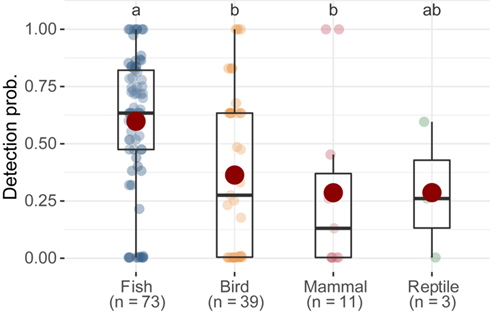
图5 不同脊椎动物类群基于eDNA的检测概率比较
本研究表明,可以利用eDNA方法对城市复杂生境中多类群生物多样性进行快速评估。在城市水体中开展基于eDNA方法的多脊椎动物类群监测具有高效及非损伤性等优点,且eDNA方法较少受生境类型和调查生物类别的影响,能在有限成本条件下为生物多样性调查提供更精细时空尺度的数据。随着该方法的不断优化和标准化,未来将能够对城市各生物类群多样性开展常规的全面调查,以及时了解不同生物群落的分布动态,发现城市生物多样性热点,从而为全球快速城市化进程中的区域生物多样性保护及生物入侵管理提供适时策略。

论文第一作者(左上)及实验室成员在进行北京地区环境水样品采集工作
北京大学生命科学学院博士后张姗为本论文的第一作者,姚蒙为论文的通讯作者。该研究得到了云南社会发展项目、国家自然基金委青年基金、青年人才托举工程、青藏二次科考项目、科技部基础资源调查项目、生科院启东产业创新基金等的资金支持。

